Epissage du pré-ARNm
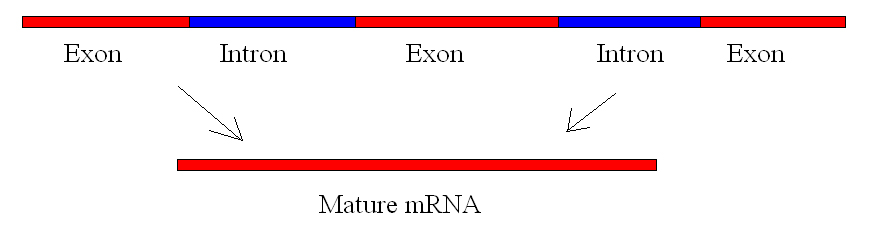
Les gènes eucaryotes sont composés d’exons, qui correspondent à des séquences codant pour des protéines (ex-on signifie qu’elles sont exprimées), et de séquences intermédiaires appelées introns (int-ron désigne leur rôle d’intervention), qui peuvent être impliquées dans la régulation du gène mais sont éliminées du pré-ARNm au cours du traitement (figure 2). Les séquences introns dans l’ARNm ne codent pas pour des protéines fonctionnelles.
La découverte des introns a surpris les chercheurs des années 1970 qui s’attendaient à ce que les pré-ARNm spécifient des séquences protéiques sans autre traitement, comme ils l’avaient observé chez les procaryotes. Les gènes des eucaryotes supérieurs contiennent très souvent un ou plusieurs introns. Ces régions peuvent correspondre à des séquences régulatrices ; cependant, la signification biologique de la présence de nombreux introns ou d’introns très longs dans un gène n’est pas claire. Il est possible que les introns ralentissent l’expression des gènes parce qu’il faut plus de temps pour transcrire les pré-ARNm contenant beaucoup d’introns. Il se peut également que les introns soient des restes de séquences non fonctionnelles issus de la fusion d’anciens gènes au cours de l’évolution. Cette hypothèse est étayée par le fait que des exons séparés codent souvent pour des sous-unités ou des domaines protéiques distincts. Dans la plupart des cas, les séquences des introns peuvent être mutées sans affecter le produit protéique.
Tous les introns d’un pré-ARNm doivent être complètement et précisément éliminés avant la synthèse des protéines. Si le processus se trompe ne serait-ce que d’un seul nucléotide, le cadre de lecture des exons rejoints se décalerait, et la protéine résultante serait dysfonctionnelle. Le processus de suppression des introns et de reconnexion des exons est appelé épissage (figures 2 et 3). Les introns sont éliminés et dégradés alors que le pré-ARNm est encore dans le noyau. L’épissage s’effectue par un mécanisme spécifique à la séquence qui garantit que les introns seront supprimés et les exons reconnectés avec l’exactitude et la précision d’un seul nucléotide. L’épissage des pré-ARNm est réalisé par des complexes de protéines et de molécules d’ARN appelés spliceosomes (figure 3).
Notez que plus de 70 introns individuels peuvent être présents, et chacun doit subir le processus d’épissage – en plus du coiffage 5′ et de l’ajout d’une queue poly-A – juste pour générer une seule molécule d’ARNm traduisible.