Empalme del pre-ARNm
Los genes eucarióticos se componen de exones, que corresponden a secuencias codificantes de proteínas (ex-on significa que se expresan), y de secuencias intermedias denominadas intrones (int-ron denota su función interventora), que pueden estar implicadas en la regulación del gen pero que se eliminan del pre-ARNm durante el procesamiento (Figura 2). Las secuencias de intrones en el ARNm no codifican proteínas funcionales.
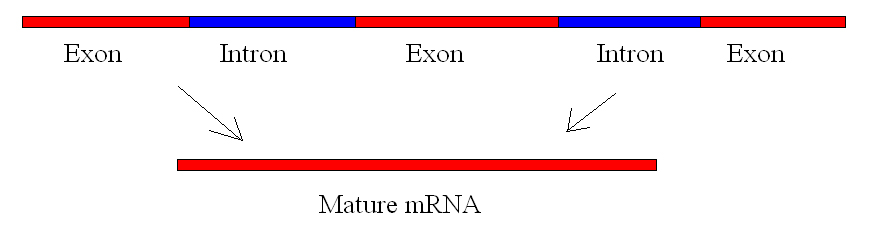
El descubrimiento de los intrones sorprendió a los investigadores de la década de 1970, que esperaban que los pre-ARNm especificaran las secuencias de las proteínas sin necesidad de procesamiento adicional, como habían observado en los procariotas. Los genes de los eucariotas superiores contienen muy a menudo uno o varios intrones. Estas regiones pueden corresponder a secuencias reguladoras; sin embargo, no está claro el significado biológico de tener muchos intrones o de tener intrones muy largos en un gen. Es posible que los intrones ralenticen la expresión del gen porque se tarda más en transcribir los pre-ARNm con muchos intrones. Otra posibilidad es que los intrones sean restos de secuencias no funcionales que han quedado de la fusión de genes antiguos a lo largo de la evolución. Esto se ve respaldado por el hecho de que los exones separados suelen codificar subunidades o dominios proteicos separados. En su mayor parte, las secuencias de los intrones pueden mutarse sin afectar en última instancia al producto proteico.
Todos los intrones de un pre-ARNm deben eliminarse de forma completa y precisa antes de la síntesis proteica. Si el proceso se equivoca incluso en un solo nucleótido, el marco de lectura de los exones reunidos se desplazaría y la proteína resultante sería disfuncional. El proceso de eliminación de intrones y reconexión de exones se denomina splicing (figuras 2 y 3). Los intrones se eliminan y se degradan mientras el pre-ARNm está todavía en el núcleo. El empalme se produce mediante un mecanismo de secuencia específica que garantiza la eliminación de los intrones y la reconexión de los exones con la exactitud y precisión de un solo nucleótido. El empalme de los pre-ARNm se lleva a cabo mediante complejos de proteínas y moléculas de ARN denominados espliceosomas (Figura 3).
Nótese que puede haber más de 70 intrones individuales, y que cada uno de ellos tiene que someterse al proceso de empalme -además del taponamiento 5′ y la adición de una cola poli-A- sólo para generar una única molécula de ARNm traducible.